ANÁLISIS MOLECULAR DE LA MICROBIOTA DUODENAL DE NIÑOS Y ADULTOS CON Y SIN ENFERMEDAD CELIACA
Esther Nistal1, Jenifer Pérez-Andrés1, Alexandra R Herrán1, Alberto Caminero4, Santiago Vivas3, José M Ruiz de Morales2, Ricardo Vicente Ullan6, y Javier Casqueiro 1,
Área de Microbiología, Facultad de Ciencias Biológicas y Ambientales, Universidad de León. Departamento de 2 Inmunología y 3 Gastroenterología, Hospital de León. 4 Instituto de Biología Molecular, Genómica y Proteómica (INBIOMIC), e Instituto de Biomedicina (IBIOMED) Campus de Vegazana, Universidad de León. 6 Instituto de Biotecnología de León. León.
RESUMEN
El gluten, un componente muy común en la dieta humana, es capaz de activar la patogénesis de la enfermedad celiaca en individuos genéticamente predispuestos. Aunque se conoce muy bien la función de las enzimas digestivas humanas sobre las proteínas del gluten, existe un gran desconocimiento sobre el papel que lleva a cabo la microbiota intestinal en el metabolismo del mismo. El objetivo de este estudio fue analizar molecularmente mediante PCR-DGGE la microbiota duodenal que participa en el metabolismo del gluten. Para ello se cultivaron biopsias duodenales de niños y adultos en un medio de cultivo (MCB) que contenía gluten como única fuente de nitrógeno.
El análisis mediante PCR-DGGE mostró que la microbiota duodenal capaz de crecer en MCB está dominada por Streptococcus salivarius y S. oralis. Sin embargo, también se detectaron otros géneros como Lactobacillus, Gemella, Haemophilus, Clostridium, Granulicatella y Staphylococcus, aunque con una frecuencia más baja. Por tanto, aunque no se pudo asociar ningún perfil electroforético a la enfermedad, sí que se ha observado una gran diversidad de bacterias duodenales que podrían estar involucradas en el metabolismo del gluten. Son necesarios más estudios que permitan caracterizar en detalle la microbiota duodenal asociada al metabolismo de gluten, con el fin de plantear en un futuro posibles alternativas terapéuticas.
INTRODUCCIÓN
La enfermedad celiaca (EC) es una enteropatía inflamatoria crónica del intestino delgado que aparece en individuos genéticamente susceptibles y es causada por una respuesta inmune inapropiada a las proteínas del gluten (1, 2). Actualmente, el único tratamiento efectivo para la EC es la eliminación total del gluten de la dieta durante toda la vida del paciente (3).
El gluten está formado principalmente por proteínas, las cuales son resistentes a la digestión completa por las enzimas digestivas humanas debido a su alto contenido en prolina y glutamina. Como consecuencia de esta digestión parcial, se originan péptidos de gran tamaño que aparecen en el lumen intestinal, los cuales generan una respuesta inmune inadecuada en pacientes con EC (4). Estudios recientes han mostrado que la microbiota intestinal desarrolla una intensa actividad metabólica en el organismo humano, actuando en la digestión de componentes de la dieta, como polisacáridos complejos y, en menor medida, proteínas, entre las que se incluye el gluten (5). Sin embargo, hay un conocimiento escaso sobre el papel que llevan a cabo los microorganismos en el metabolismo del gluten. Estudios recientes han mostrado que la microbiota intestinal puede ejercer un papel fundamental en la digestión de las proteínas del gluten y de los péptidos derivados. Aunque se considera que la digestión de los compuestos proteicos comienza en el estómago, se ha descrito una actividad proteolítica de origen bacteriano en la cavidad oral. Esta actividad es capaz de hidrolizar péptidos ricos en prolina como los que aparecen en las proteínas del gluten (6). Dentro de las bacterias orales que generan esta actividad se han identificado Rothia mucilaginosa y R. aeria, las cuales son capaces de digerir eficientemente estas proteínas (7). Curiosamente, especies de Rothia han sido también descritas en el duodeno (8). Otras bacterias relacionadas con el metabolismo del gluten en la cavidad oral son Streptococcus mitis, S. pneumoniae, Staphylococcus epidermidis, Bifidobacterium longum y B. dentium (7). A esta idea se suma un estudio realizado por nuestro grupo, que ha descrito una actividad glutenásica fecal (AGF) relacionada directamente con la ingesta de gluten; dicha actividad podría ser derivada del metabolismo microbiano en el intestino (5). Otros estudios muestran que cepas de Bifidobacterium y Bacteroides fragilis, aisladas de las heces humanas, son capaces de digerir péptidos derivados del gluten (9, 10). Caminero et al (11)mostraron una amplia variedad de microorganismos pertenecientes a la microbiota fecal capaces de utilizar el gluten como nutriente, lo que apunta claramente a la gran importancia de la microbiota en el metabolismo del gluten. En consecuencia de este hecho y de acuerdo con las nuevas evidencias que señalan a una posible contribución bacteriana en el desarrollo de la EC (12, 13, 14, 15), nos planteamos estudiar molecularmente, mediante PCR-DGGE, la microbiota duodenal que participa en el metabolismo del gluten a partir del cultivo de biopsias de niños y adultos en un medio que contiene gluten como única fuente de nitrógeno.
MATERIALES Y MÉTODOS
Pacientes y Muestras duodenales
Se recogieron biopsias duodenales de forma consecutiva de niños (rango de edad 1-12 años) y adultos (rango de edad entre 15-40 años) procedentes de los Servicios de Gastroenterología de adultos y pediátrica del Hospital de León que acudieron a la consulta con síntomas. 24 sujetos fueron incluidos en este estudio: 8 individuos adultos no enfermos celiacos (No-ECA) (4 hombres y 4 mujeres), 3 pacientes adultos enfermos celiacos activos (ECAA) (2 hombres y 1 mujer), 5 adultos enfermos celiacos tratados (ECTA) (2 mujeres y 3 hombres), 2 niños no enfermos celiacos (No-ECN) (1 niño y 1 niña) y 6 niños enfermos celiacos activos (ECAN) (4 niños y 2 niñas). Al diagnóstico, todos los pacientes celiacos presentaban datos clínicos compatibles con la enfermedad, como serología positiva (anticuerpos transglutaminasa), genética positiva (HLA-DQ2-DQ8), y alteraciones histológicas en la mucosa de la biopsia duodenal. Las muestras de los pacientes ECA fueron recogidas en el momento del diagnóstico antes de comenzar la dieta sin gluten. Los pacientes ECT en el momento de la toma de la biopsia llevaban más de un año en dieta sin gluten. En el caso de los individuos No-EC, las muestras de biopsias duodenales fueron obtenidas de pacientes que fueron referidos a las consultas de gastroenterología debido a otras patologías intestinales (diarrea crónica, hernia de hiato, etc.). Ninguno de los individuos incluidos en el estudio fue tratado con antibióticos durante al menos un mes antes de la toma de las muestras. Las biopsias fueron recogidas mediante pinza de biopsia convencional a través del canal de trabajo de un videogastrocopio. En el caso de los niños se utilizó un fibrogastroscopio pediátrico con una pinza de menor tamaño adaptada para el canal de trabajo. En los niños se utilizó siempre sedación profunda que practicó un pediatra entrenado en la UCI pediátrica. En los adultos se ofrecía sedación profunda con propofol que controlaba el propio endoscopista que tomaba la biopsia. En total se tomaban 2 biopsias en los adultos y 3 en los niños de la segunda y tercera porción duodenal. Estas biopsias eran las primeras en tomarse, antes de las enviadas para anatomía patológica, y evitar así contaminaciones. Según eran tomadas las biopsias, se inoculaba directamente en un tubo de ensayo con 1 cc de suero salino. Estas muestras eran procesadas de forma inmediata a temperatura ambiente. En todos los casos se obtuvo el consentimiento informado tanto de los padres de los niños, como de los pacientes adultos incluidos en el estudio a la toma de muestras. Los protocolos de estudio fueron aprobados por la comisión de ética del Hospital de León. Todas las muestras fueron extraídas mediante endoscopia alta con biopsia de la 2ª-3ª porción duodenal y fueron procesadas de inmediato.
Medio de cultivo y condiciones de cultivo
Para el aislamiento de los microorganismos a partir del cultivo de biopsias se empleó el medio líquido MCB diseñado en nuestro laboratorio que contiene gluten como única fuente de nitrógeno: CaCl2 2mM, ZnSO4 2mM, Glucosa 2%, Tween 80 0.1% (v/v), FeCl3 20µM, Gluten (Sigma-Aldrich) 3%, y digerido de gluten con pepsina 0.5%. El pH fue ajustado a 6,5 con un tampón fosfato (1%). El medio se esterilizó en el autoclave durante 20 minutos a 121ºC. Seguidamente mediante cultivo en placa se comprobó que el medio estaba totalmente estéril.
El digerido de gluten al 3% (100 mL) se obtuvo al incubar 3 g de gluten con 1 g de pepsina a pH 2 ajustado con HCl (10M). La mezcla se incubó durante 2 horas a 37ºC con agitación (250 rpm). Seguidamente se elevó el pH a 6,5 con NaOH (1M) y posteriormente se esterilizó a 121ºC en el autoclave durante 20 minutos.
La biopsia se inoculó en un matraz con 10 mL de medio de cultivo y se incubó durante 48 horas a 37ºC con agitación (100 rpm) en condiciones microóxicas.
Extracción de ADN de los cultivos
de biopsia y amplificación por PCR
Las extracciones de ADN genómico de los cultivos de biopsia se llevó a cabo siguiendo las instrucciones del protocolo del kit SpeedTools Tissue DNA Extraction (Biotools, España). La concentración de ADN fue determinada empleando el espectrofotómetro NanoDrop ND-1000 (Saveen&Werner, Limhann, Suecia).
Para caracterizar molecularmente la microbiota duodenal se amplificó mediante PCR dos regiones variables de diferente tamaño del ADN ribosomal 16S y así poder comparar los resultados obtenidos con ambos fragmentos. Para la amplificación de los genes del ARNr 16S ello se empleóaron dos parejas de cebadores. la pareja de oligonucleótidos HDA1GC y HDA2 (16) que amplifican un fragmento de unos 200 pb correspondientes a la región V3 del ADNr 16S y la pareja de cebadores F968GC y R1401 (17) que amplifican un fragmento de aproximadamente 450 pb de la región V6-V8 del ADNr 16S. Para dichas amplificaciones se usó el kit comercial iProof High-Fidelity Master Mix (BioRad). El programa de PCR que se utilizó para este kit consistió en una desnaturalización inicial de 98ºC durante 30 segundos y a partir de aquí 30 ciclos con las siguientes temperaturas: 98ºC durante 10 segundos, 56ºC ó 55ºC durante 30 segundos y 72ºC, 30 segundos. El programa de PCR finalizó con una extensión a 72ºC durante 10 minutos.
Análisis de la microbiota obtenida a partir del cultivo de biopsias duodenales mediante electroforesis en gradiente desnaturalizante (DGGE)
Los productos de PCR de la región V3 y V6-V8 del ADNr 16S fueron separados mediante la técnica del DGGE en geles del 10% y 8% de acrilamida/bisacrilamida en un gradiente desnaturalizante de urea (7M) y formamida (40% v/v) del 35-55% y 40-60% respectivamente. El análisis de DGGE de los productos de PCR se realizó en el aparato DCode Universal Mutation Detection System (Bio-Rad, Richmond, CA), a un voltaje constante de 70 V y una temperatura de 60ºC durante 15 horas. Los geles fueron teñidos con bromuro de etidio durante 30 minutos y visualizados bajo un transiluminador con luz ultravioleta. Las bandas de interés fueron cortadas del gel de poliacrilamida con un cubre estéril bajo luz ultravioleta e incubadas en 20 µL de agua milliQ estéril a 4ºC durante toda la noche. Alícuotas de 5 µL fueron empleadas como molde en una nueva reacción de PCR. Se llevó a cabo la reamplificación de la región ribosomal con el par de oligonucleótidos correspondiente pero sin portar la cola de poli-GC. Los fragmentos reamplificados fueron purificados y clonados con el sistema StrataClone PCR Cloning (Stratagene, La Jolla, CA) para posteriormente ser secuenciados en el Servicio de secuenciación de la Universidad de León y estas se compararon con las secuencias depositadas en la base de datos de GeneBank con el algoritmo del BLASTn. Para completar la identificación molecular de estas secuencias, se llevó a cabo un análisis filogenético. Las secuencias con un porcentaje ≥ 97% de identidad con respecto a las secuencias de bacterias conocidas después del análisis filogenético fueron consideradas de la misma especie.
Análisis estadístico
Las diferencias en número de especies entre los distintos grupos de poblaciones fueron analizadas empleando el test “chi-cuadrado” (se aplicó la corrección de Yates). La significación estadística fue establecida para valores de p<0.05.
RESULTADOS
Análisis molecular mediante PCR-DGGE
de la región V3 del ADNr 16S
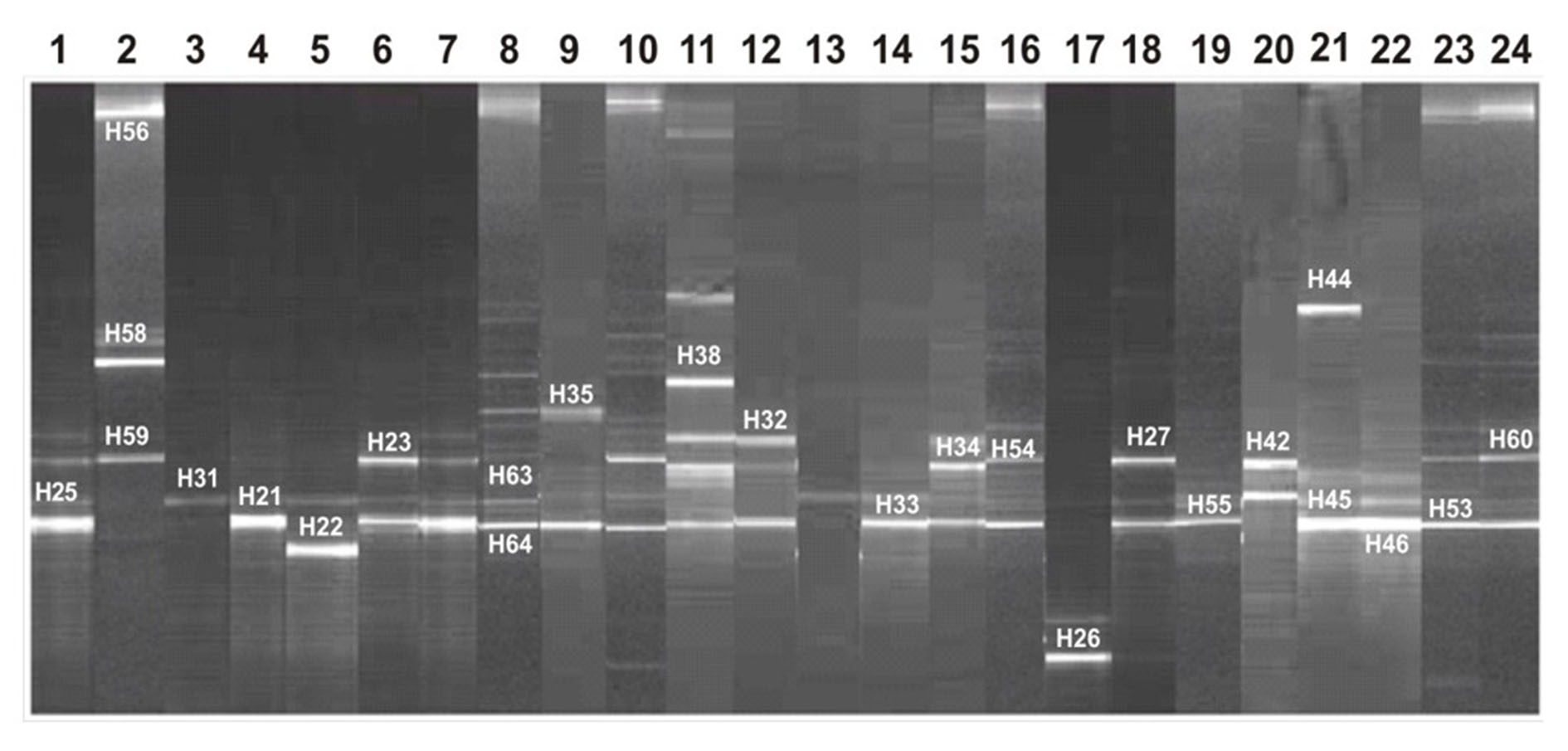
En la Figura 1 se observan los perfiles de DGGE obtenidos a partir de los productos de PCR de la región V3 empleando como molde el ADN de los cultivos de biopsias duodenales. Los diferentes perfiles mostraron entre 1 a 5 bandas intensas y bien resueltas. El resto de las bandas eran tenues o daban lugar a un smear. No se observaron diferencias estadísticamente significativas en cuanto al número de bandas entre los perfiles electroforéticos de individuos sanos y de enfermos celiacos, tanto activos como tratados, ni tampoco se apreciaron diferencias asociadas a la edad.
Se seleccionaron varias bandas para llevar a cabo su identificación molecular. Los productos de PCR que fueron identificados por secuenciación a partir de las bandas del DGGE aparecen en la Tabla I. Para completar la identificación molecular de cada una de las bandas secuenciadas se llevó a cabo un análisis filogenético. La frecuencia de las diferentes especies detectadas mediante PCR-DGGE se muestra en la Tabla II. Dentro de las diferentes especies identificadas, predominó Streptococcus salivarius y S. oralis que se detectaron en prácticamente todos los individuos, tanto niños como adultos independientemente de la enfermedad. Por el contrario, Bacillus cereus, Haemophilus influenza, Lactobacillus mucosae y Staphylococcus epidermidis sólo fueron detectados a partir del cultivo de biopsias de niños, pero con una frecuencia muy baja. A partir del cultivo de biopsias procedentes de individuos adultos, también aparecieron con baja frecuencia especies como Clostridium perfringens, Granulicatella elegans o G. adiacens, Lactobacillus crispatus y L. gasseri.
Análisis molecular mediante PCR-DGGE de la región V6-V8 del ADNr 16S
Los productos de PCR generados por los cebadores F968GC/R1401 fueron separados mediante DGGE en geles del 8% de acrilamida/bisacrilamida en un gradiente desnaturalizante del 40-60% de urea y formamida. Como se observa en la Figura 2, los patrones de bandas de DGGE obtenidos con los cebadores universales correspondientes a la región V6-V8 del ADNr 16S, mostraron muy pocas bandas (entre 1 a 4 bandas), pero la gran mayoría estaban bien resueltas. No se pudo asociar ningún perfil electroforético a la enfermedad. Tampoco se observaron diferencias en cuanto a número de bandas presentes en los perfiles electroforéticos de niños y de adultos, ni entre sanos y enfermos.
A partir de los geles, se seleccionaron varias bandas para llevar a cabo su identificación molecular. Los productos de PCR que fueron identificados por secuenciación a partir de las bandas del DGGE aparecen en la Tabla III. La frecuencia de cada una de esas bandas se muestra en la Tabla IV. Los resultados fueron similares a los obtenidos con los cebadores que amplifican la región V3, ya que las especies identificadas fueron las mismas, con la aparición de algunas nuevas. De nuevo, los fragmentos amplificados correspondientes a las especies Streptococcus oralis y S. salivarius aparecieron en prácticamente todos los individuos sanos y enfermos, independientemente de la edad. Por el contrarioGranulicatella elegans o G. adiacens fue detectada en 4 de los 6 niños ECA y no aparece en ninguno de los niños sanos. El resto de las especies identificadas presentaban una frecuencia muy baja, ya que aparecían en tan solo una o dos de las biopsias analizadas (Tabla IV).

Figura 1.- Perfiles de DGGE de las comunidades de bacterias obtenidas a partir del cultivo en un medio con gluten de biopsias procedentes de niños no enfermos celiacos (No-ECN) (carriles 1 y 2), niños enfermos celiacos activos (ECAN) (carriles desde el 3 al 8), adultos no enfermos celiacos (No-ECA) (carriles del 9 al 16), adultos enfermos celiacos activos (ECAA) (carriles del 17 al 19) y adultos enfermos celiacos tratados (ECTA) (carriles del 20 al 24). Los cebadores empleados para llevar a cabo la reacción de PCR fueron HDA1GC/HDA2. Los números indican los fragmentos secuenciados, cuya identificación se muestra en la Tabla I



Figura 2.- Perfiles de DGGE de las comunidades de bacterias obtenidas a partir del cultivo de biopsias en un medio con gluten de niños no enfermos
celiacos (No-ECN) (carriles 1 y 2), niños enfermos celiacos activos (ECAN) (carriles del 3 al 8), adultos no enfermos celiacos (No-ECA)
(carriles del 9 al 16), adultos enfermos celiacos activos (ECAA) (carriles del 17 al 19) y adultos enfermos celiacos tratados (ECTA) (carriles del
20 al 24). Los cebadores empleados para llevar a cabo la reacción de PCR fueron F968GC/R1401. Los números indican los fragmentos secuenciados,
cuya identificación se muestra en la Tabla III

DISCUSIÓN
El conocimiento de las bacterias duodenales involucradas en el metabolismo del gluten es escaso. Es importante estudiar qué bacterias participan y cómo participan puesto que las proteínas del gluten son responsables de la activación de la patogénesis de la EC. Para el estudio de estas bacterias duodenales se ha empleado un medio de cultivo diseñado en nuestro laboratorio, el MCB, que lleva gluten como única fuente de nitrógeno, lo que va a permitir la proliferación de bacterias capaces de emplear las proteínas del gluten como nutriente. El análisis mediante PCR-DGGE mostró que la microbiota duodenal capaz de crecer en el MCB está dominada por Streptococcus salivarius y S. oralis, ya que fueron identificados en el 71% y 50% (respectivamente) de los cultivos de biopsias analizados. La predominancia del género Streptococcus en el duodeno ha sido reflejada previamente en varios trabajos que han empleado técnicas clásicas de cultivo para identificar la microbiota duodenal (18, 19). Además, estudios de caracterización de la microbiota intestinal involucrada en el metabolismo del gluten en la cavidad oral y el intestino grueso han mostrado la presencia de cepas de Streptococcus capaces de hidrolizar el gluten (11, 20, 21). Por tanto, estas bacterias duodenales podrían jugar un papel importante en el metabolismo del gluten, participando en la hidrólisis bacteriana de péptidos inmunogénicos del gluten en el intestino delgado. En los últimos años, varias bacterias capaces de digerir gluten han sido propuestas como posible tratamiento de la EC (21, 22); sin embargo ninguna de ellas es miembro hospedador de la microbiota duodenal, lo que dificulta su uso clínico. Así mismo, diferentes estudios han descrito que determinadas cepas de Streptococcus salivariuspueden ser empleadas como probióticos, ya que son capaces de inducir una respuesta anti-inflamatoria y ejercer un efecto protector frente a la apoptosis inducida por patógenos (23, 24).
Otros géneros como Lactobacillus, Gemella, Haemophilus, Clostridium, Granulicatella y Staphylococcus también se detectan mediante PCR-DGGE, aunque con una frecuencia más baja. Lo que caracteriza a todos estos microorganismos, es que son capaces de crecer sobre un medio de cultivo que contiene el gluten como única fuente de nitrógeno. Esto puede ser explicado por diferentes razones; i) presentan actividad proteolítica frente al gluten; ii) por el contrario carecen de esa actividad, pero son capaces de crecer en ese medio gracias a los nutrientes que le aportan otros microorganismos que si son capaces de hidrolizar el gluten; iii) o su crecimiento se ve favorecido, porque son capaces de emplear los péptidos de menor tamaño que se originan como consecuencia de la digestión parcial del gluten con pepsina. Estudios previos han mostrado distintas cepas bacterianas de géneros como Clostridium, Lactobacillus y Staphylococcus son capaces de hidrolizar gluten (11). Por tanto, estos microorganismos pueden tener un papel en el metabolismo del gluten en el duodeno.
Varios trabajos han mostrado una disbiosis intestinal en pacientes con EC (12-15). Sin embargo, los mecanismos mediante los cuales las bacterias pueden participar en la patogénesis se desconocen. Se ha mostrado que algunos de los grupos bacterianos alterados en la EC están relacionados con el metabolismo del gluten (11, 13, 14, 25, 26). Así mismo, Bernardo et al, (27) mostraron la existencia de un patrón específico de metaloproteasas bacterianas con capacidad de degradar gliadina en la mucosa duodenal de pacientes con EC diferente al patrón presente en los sanos. Nuestro trabajo teniendo en cuenta el número reducido de pacientes analizados de cada tipo, no muestra un perfil de especies bacterianas asociadas a la EC o a los individuos no enfermos celiacos. Quizás las diferencias podrían estar asociadas a la funcionalidad de las distintas cepas que son capaces de crecer en el medio MCB. Los enterotipos que determinan la microbiota intestinal, en la mayoría de los casos se definen por la composición de especies. Pero las funciones moleculares no siempre son llevadas a cabo por las especies más abundantes. Por ello resulta necesario llevar a cabo también un análisis funcional para poder entender el papel de las comunidades microbianas (28). Además, la técnica PCR-DGGE es capaz de detectar entre el 90-99% de las especies más representativas de una comunidad, sin discriminar entre células vivas, células muertas o células en un estado no cultivable. Sin embargo, esta técnica no refleja la presencia de otras especies de bacterias menos representativas que si pueden ser detectadas mediante colecciones de clones o mediante técnicas clásicas de cultivo (29, 30).
La baja diversidad bacteriana obtenida con el medio MCB puede deberse a las condiciones adversas que presenta el duodeno como por ejemplo pH ácido, peristaltismo o la presencia de ácidos biliares. Además, se ha utilizado un medio de cultivo de enriquecimiento con gluten, el cual se caracteriza por presentar secuencias peptídicas de difícil digestión. Por estos motivos, es posible que una parte de los microorganismos presentes en el duodeno no puedan crecer en el medio de cultivo MCB. Nistal et al, (8) mostraron que una parte importante de la microbiota duodenal de los adultos está formada por bacterias aún no cultivadas. A menudo, para el cultivo de microorganismos del tracto gastrointestinal se emplean medios de cultivo complejos (como por ejemplo Brain Heart Infusion broth, Brucella broth, etc.,) (31, 32) que se caracterizan por ser medios ricos en nutrientes ya que en su composición se incluyen hidrolizados de productos animales o vegetales, como caseína, carne, soja, extracto de levaduras u otras sustancias nutritivas que favorecen el crecimiento de muchos microorganismos. Sin embargo, el medio MCB carece de esos componentes, lo que hace que se trate de un medio de cultivo más exigente para el crecimiento de los microorganismos. Esto permite la proliferación y caracterización de bacterias que directa o indirectamente están involucradas en el metabolismo del gluten.

RECONOCIMIENTOS
Este trabajo ha sido financiado por un proyecto del Instituto de Salud Carlos III, Fondo de Investigación Sanitaria cofinanciado por FEDER (FIS P110/02447) y por un proyecto de la Junta de Castilla y León, Consejería de Sanidad (Ref. 520/A/10). Esther Nistal y Alexandra Rodríguez recibieron una beca de la Junta de Castilla y León, cofinanciada por el Fondo Social Europeo. Jénifer Pérez-Andrés recibió una beca del Ministerio de Educación, Cultura y Deporte del Gobierno de España
BIBLIOGRAFÍA
1. Ludvigsson JF, Leffler DA, Bai JC, Biagi F, Fasano A, Green PHR, et al. The Oslo definitions for coeliac disease and related terms. Gut 2012;62(1):43-52.
2. Hill ID, Dirks MH, Liptak GS, Colletti RB, Fasano A, Guandalini S, et al. Guideline for the diagnosis and treatment of celiac disease in children: recommendations of the North American Society for Pediatric Gastroenterology, Hepatology and Nutrition. J Pediatr Gastroenterol Nutr 2005;40(1):1-19.
3. Ciclitira PJ, Johnson MW, Dewar DH, Ellis HJ. The pathogenesis of coeliac disease. Mol Aspects Med 2005; 26: 421-58.
4. Shan L, Molberg Ø, Parrot I, Hausch F, Filiz F, Gray GM, et al. Structural basis for gluten intolerance in celiac sprue. Science 2002; 297: 2275-9.
5. Caminero A, Nistal E, Arias L, Vivas S, Comino I, Real A, et al. A gluten metabolism study in healthy individuals shows the presence of faecal glutenasic activity. Eur J Nutr, 2012; 51(3): 293-9.
6. Helmerhorst EJ, Zamakhchari M, Schuppan D, Oppenheim FG. Discovery of a novel and rich source of gluten-degrading microbial enzymes in the oral cavity. PLoS One 2010; 5(10): e13264.
7. Zamakhchari M, Wei G, Dewhirst F, Lee J, Schuppan D, Oppenheim FG, et al. Identification of Rothia bacteria as gluten-degrading natural colonizers of the upper gastro-intestinal tract. PLoS One 2011; 6(9): e24455.
8. Nistal E, Caminero A, Herrán AR, Arias L, Vivas S, de Morales JM et al. Differences of small intestinal bacteria populations in adults and children with/without celiac disease: effect of age, gluten diet, and disease. Inflamm Bowel Dis 2012; 18(4): 649-56.
9. Laparra JM, Sanz Y. Bifidobacteria inhibit the inflammatory response induced by gliadins in intestinal epithelial cells via modifications of toxic peptide generation during digestion. J Cell Biochem 2010; 109(4): 801-7.
10. Sánchez E, Laparra JM, Sanz Y. Discerning the Role of Bacteroides fragilis in Celiac Disease Pathogenesis. Appl Environ Microbiol 2012; 78(18): 6507-15.
11. Caminero A, Herrán AR, Nistal E, Pérez-Andrés J, Vaquero L, Vivas S, et al. Diversity of the cultivable human gut microbiome involved in gluten metabolism: isolation of microorganisms with potential interest for coeliac disease. FEMS Microbiol Ecol 2014; 88: 309-19.
12. Forsberg G, Fahlgren A, Horstedt P, Hammarstrom S, Hernell O, Hammarstrom ML. Presence of bacteria and innate immunity of intestinal epithelium in childhood celiac disease. Am J Gastroenterol 2004; 99: 894-904.
13. Collado MC, Calabuig M, Sanz Y. Differences between the fecal microbiota of coeliac infants and healthy controls. Curr Issues Intest Microbiol 2007; 8: 9-14.
14. Nadal I, Donat E, Ribes-Koninckx C, Calabuig M, Sanz Y. Imbalance in the composition of the duodenal microbiota of children with coeliac disease. J Med Microbiol 2007; 56: 1669-74.
15. Sanz Y, Nadal I, Sánchez E. Probiotics as drugs against human gastrointestinal infections. Recent Pat Antiinfect Drug Discov 2007;2:148-56.
16. Walter. J, Tannock GW, Tilsala-Timisjarvi A, Rodtong S, Loach DM, Munro K, et al. Detection and identification of gastrointestinal Lactobacillus species by using denaturing gradient gel electrophoresis and species-specific PCR primers. Appl Environ Microbiol 2000;66:297-303.
17. Matsuki T, Watanabe K, Tanaka R, Oyaizu H. Rapid identification of human intestinal bifidobacteria by 16S rRNA-targeted species- and group-specific primers. FEMS Microbiol Lett 1998;167:113-21.
18. Sullivan A, Tornblom H, Lindberg G, Hammarlund B, Palmgren AC, Einarsson C, et al. The micro-flora of the small bowel in health and disease. Anaerobe 2003;9:11-4.
19. Zilberstein B, Quintanilha AG, Santos MA, Pajecki D, Moura EG, Alves PR, et al. Digestive tract microbiota in healthy volunteers. Clinics (Sao Paulo) 2007;62:47-54.
20. Fernandez-Feo M, Wei G, Blumenkranz G, Dewhirst FE, Schuppan D, Oppenheim FG, et al. The cultivable human oral gluten-degrading microbiome and its potential implications in coeliac disease and gluten sensitivity. Clin Microbiol Infect 2013;19:E386-94.
21. Tian N, Wei G, Schuppan D, Helmerhorst EJ. Effect of Rothia mucilaginosa enzymes on gliadin (gluten) structure, deamination, and immunogenic epitopes relevant to celiac disease. Am J Physiol Gastrointest Liver Physiol 2014;307:G769-76.
22. Laparra JM, Olivares M, Gallina O, Sanz Y. Bifidobacterium longum CECT 7347 modulates immune responses in a gliadin-induced enteropathy animal model. PloS One 2012;7: e30744.
23. Cosseau C, Devine DA, Dullaghan E, Gardy JL, Chikatamarla A, Gellatly S, et al. The commensal Streptococcus salivarius K12 downregulates the innate immune responses of human epithelial cells and promotes host-microbe homeostasis. Infect Immun 2008;76:4163-75.
24. Sliepen I, Van Damme J, Van Essche M, Loozen G, Quiryne M, Teughels W. Microbial interactions influence inflammatory host cell responses. J Dent Res 2009;88:1026-30.
25. Sanz Y, Sánchez E, Marzotto M, Calabuig M, Torriani S, Dellaglio F. Differences in faecal bacterial communities in coeliac and healthy children as detected by PCR and denaturing gradient gel electrophoresis. FEMS Immunol Med Microbio 2007;51:562-8.
26. Collado MC, Donat E, Ribes-Koninckx C, Calabuig M, Sanz Y. Specific duodenal and faecal bacterial groups associated with paediatric coeliac disease. J Clin Pathol 2009;62:264-9.
27. Bernardo D, Garrote JA, Nadal I, León AJ, Calvo C, Fernández-Salazar L, et al. Is it true that coeliacs do not digest gliadin? Degradation pattern of gliadin in coeliac disease small intestinal mucosa. Gut 2009;58:886-7.
28. Arumugam M, Raes J, Pelletier E, Le Paslier D, Yamada T, Mende DR, et al. Enterotypes of the human gut microbiome. Nature 2011;473:174-80.
29. Cocolin L, Manzano M, Cantoni C, Comi G. Denaturing gradient gel electrophoresis analysis of the 16S rRNA gene V1 region to monitor dynamic changes in the bacterial population during fermentation of Italian sausages. Appl Environ Microbiol 2001;67:5113-21.
30. Van Beek S, Priest FG. Evolution of the lactic acid bacterial community during malt whisky fermentation: a polyphasic study. Appl Environ Microbiol 2002;68:297-305.
31. Yun JH, Yim DS, Kang JY, Kang BY, Shin EA, Chung MJ, et al. Identification of Lactobacillus ruminus SPM0211 isolated from healthy Koreans and its antimicrobial activity against some pathogens. Arch Pharm Res 2008;28:660-6
32. Sainsus N, Cattori V, Lepadatu C, Hofmann-Lehmann R. Liquid culture medium for the rapid cultivation of Helicobacter pylori from biopsy specimens. Eur J Clin Microbiol Infect Dis 2008; 27:1209-17.
CORRESPONDENCIA:
Esther Nistal
Área de Microbiología,
Facultad de Ciencias Biológicas y Ambientales.
Universidad de León.
Telf: 987 291 504;
esthernistal@hotmail.com
